r语言实用技术(R语言学习笔记来源网络)
向量索引
- 正(负)整数索引
可以使用Length()访问向量的个数,访问向量x的第一个值可以使用x[1];还可以使用负整数进行索引,表示访问除了这一行的其他行数据,比如不访问一个三行数据的第二行,可以使用x[-2]
2、逻辑向量索引

以上代码是只输出对应逻辑值为真的值。
还可以进行逻辑判断,例如:
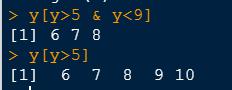
对于字符串向量,我们可以使用一些特殊的操作符进行逻辑判断,例如“%in%” ,判断前面一个向量内的元素是否在后面一个向量中,返回布尔值。

也可以用于索引:

3、名称索引
我们可以使用names()函数为向量的每一个元素添加名称:

然后即可以通过每一个元素的names来访问它的值:

添加向量:
可以直接通过索引来添加向量;

也可以一次性添加多个元素:

在向量中插入一个新元素:
可以使用append()函数,以下代码表示,在x这个向量的5这个元素后面插入一个99的元素:

如果after=0则代表在向量的头部插入数据:

删除向量:
如果想删除整个向量,可以直接使用rm()函数,如果想删除某个函数,可以直接采用负整数索引的方式:

其实就是重新生成一个新的向量,替换掉原来的向量
修改向量值:直接将需要修改的值索引出来,然后给它赋一个新的值就可以了。

但是注意这里是数值型的向量,我们不能赋值给字符串,会把整个向量变成字符型向量

向量运算
向量是R中最基本的数据结构。
向量运算是对应位置的元素进行运算,其中长的向量的个数必须是短向量个数的整数倍。Ceiling()不小于x的最小整数,floor()函数不大于x的最大整数:

Trunc()函数返回整数部分:

Round()函数用于进行四舍五入,digits用来表示返回的位数

Signif()保留小数部分有效数字

Which()函数可以返回索引值,也就是元素所在的位置

矩阵与数组
矩阵是一个按照长方阵列排列的复数或实数集合,向量是一维的,矩阵是二维的,需要有行和列。在R软件中,矩阵是有维数的向量,这里的矩阵元素可以是数值型,字符性改革或者是逻辑型,但是每个元素必须都拥有相同的模式,这个和向量一致。
我们可以通过matrix()函数来创建矩阵

行数和列数的分配要满足分配的条件,如果只给出一个行或者是一个列,R会自动进行分配,可以通过byrow来指定按行还是按列进行排列:

Dinames参数可以通过一个列表,指定矩阵行和列的名字:

Dim()函数可以显示向量的维数,可以通过dim()函数来对向量添加维数,从而构建矩阵

下面介绍一下数组这个数据结构。R中的数组其实就是多维的矩阵,我们重新定义一个向量x:

只要向dim()函数传入三个参数,就可以构建三位数组,数组还可以使用array()函数来创建:

还可以创建字符型和逻辑型的数组,那么创建了矩阵,要如何访问矩阵的数据呢,下面是矩阵的索引:
首先可以通过矩阵下标进行访问,m[1,2]表示访问第一行第二列的元素

也可以一次访问多个元素,比如访问第一行的第二、三、四列元素,访问第二、三行的第一列元素。

输出矩阵的一个子集:

如果下标只写一个数字,就是单独访问行或者列:

也可以同样使用负索引进行访问,还可以输入对应的行列名称进行访问:

矩阵中的四则运算需要行和列一致,与向量一致,可以使用colsums()、rowsums()、rowmeans()等函数对整个矩阵进行计算也可以进行矩阵的乘法:

分别是矩阵的内积(对应元素相乘)以及矩阵的外积
列表
State.center就是一个典型的列表,是美国每个州的经纬度,可以使用list()函数来创建列表:


这样就生成了一个列表,我们也可以为每个变量添加一个名称,例如:

列表中的元素不存在顺序,使用名称就可以访问数据,下面介绍一下数据列表的访问
第一种方法是可以使用索引的方式进行访问

第二种方法是可以使用名字来进行访问

另外,列表比之后的数据框多了一种$的访问方式:

对于列表还有一种双中括号的访问方式:

两者的差别在于,一个中括号其实输出的是列表的一个子集,它其实还是一个列表,因为如果一个访问多个元素,这些元素的数据类型又不同,那么输出结果只能是列表,当我们使用两个中括号进行输出,那么就是输出数据本身的类型,可以使用class()函数来测试一下

两个中括号每次只能访问一个元素,如果要像列表中添加元素,可以使用双中括号进行添加:

如果想删除列表中的元素可以使用负索引的方式,然后再赋值给原来的列表:

或者是使用Null来赋值

数据框
数据框是一种表格式的数据结构,数据框子在模拟数据集,与其他统计软件例如SAS或者SPSS中的数据集的概念一致,数据集通常是由数据构成的一个矩形数组,行表示观测,列表示变量,不同的行业对于数据集的行和列叫法不同。
数据框实际上是一个列表,列表中的元素是向量,这些向量构成数据框的列,每一列必须具有相同的长度,所以数据框是矩形结构,而且数据框的列必须命名。
矩阵和数据框的不同,矩阵必须是同一数据类型,数据框每一列必须为同一类型,每一行可以不同
数据框可以通过data.frame()函数进行创建
数据框的访问:
直接使用索引
直接使用名称进行索引
可以使用attach()将数据集存进R的内存,这样就便于访问

使用完之后可以使用detach()函数取消加载。
数据框也可以使用双中括号的方式进行访问,返回的是向量而不是列表。

因子
在R中变量可以分为名义型变量、有序型变量、连续型变量,名义型变量没有顺序的区别,有序型变量介于二者之间,不同值之间有顺序关系。
在R中,名义型变量和有序型变量被称为因子,factor,这些分类变量的可能只被称为一个水平,level,例如good、better、best,都被称为一个level,由这些水平值构成的向量就称为因子。在很多会吐函数中,输入的数据也必须是因子类型。
例如mtcars数据集,cyl这一列可以作为因子,而4、6、8就是这一列因子的水平:

那么如何来定义一个因子数据呢?
可以使用factor函数:

我们还可以在定义因子的时候人为指定level的水平:

还可以使用factor()函数将向量转变成因子:

R中有一个cut()函数,可以将连续型变量x分割成连续水平的因子:

缺失数据
缺失数据的分类:

在R中,用na代表缺失值,NA是不可用的意思,用于存储缺失信息,这里缺失值NA表示没有,但注意不一定就是0,NA是不知道是多少,也可能是0,也可能是任何值,缺失值和值为零是完全不同的。
但是有时候NA值的缺失会导致数据计算出现问题:

所以此时我们需要剔除NA值,可以通过一些函数的na.rm选项参数剔除:

但是计算平均值时是以50个总数来计算还是49个总数来计算呢,我们来验证一下这个问题:

说明是将NA值剔除之后求平均值。
可以在前期数据处理的时候就检查数据集是否存在缺失值,来进行逻辑测试,我们来测试一下VIM包中的sleep数据集:

使用is.na()函数来测试一下数据集:

可以使用colsums()和rowsums()来计算每一行的缺失值数目
如果想去除掉数据集中的缺失值,形成一个新的函数,则可以使用na.omit()函数:

使用na.omit()函数处理数据框,通常是直接删除缺失的行或者列:

但是这样处理有一个问题,就是当缺失值超过一半的时候,会对分析结果造成很大的影响,所以R中有很多处理缺失值的办法:

其他缺失数据:
缺失数据NaN,代表不可能的值
Inf表示无穷,分为正无穷Inf和负无穷Inf,代表无穷大或者无穷小。
字符串
nchar()函数可以用来统计字符串的长度:

Length()返回向量中元素的个数,而nchar返回每个元素字符串的个数:

Paste()函数用于粘贴字符串,将多个字符串合并为一个,默认使用空格分割,也可以通过sep选项参数来设置分隔符:


向量与字符串的连接是向量和字符串分别连接,例如:

Substr()函数用于提取字符串,函数的参数分别是一个原始的字符串,一个起始点和一个结束点,返回值是起始点和结束点之间的字符串。

然后使用toupper()函数便可以将单词大写,tolower()可以转换为小写。
Grep()函数可以用于查找字符串:

表示与第二个位置上的字符串匹配上了,如果fixed参数为F,则表示支持正则表达式,那么‘A ’表示匹配一到正无穷个字符A,那么“AC”也会入选。
Match()函数可以进行字符串匹配
Strsplit()可进行字符串的分割,这个函数需要两个参数,字符串和分割符:

但是这个函数返回的是一个列表,而不是向量。
日期与时间
时间序列分析:
对时间序列的描述
利用前面的结果进行预测

“ts”是time series的简称,代表时间序列数据。
在R中,日期数据别单独归为一个date类,我们可以使用sys.date()函数查看当前系统的时间:

在R中可以使用as.date()函数将数据转换为日期数据,使用format选项参数决定外观,。比如哪部分作为年,哪部分作为月

也可以使用seq()函数创建连续的时间点:

要使用as.date()系统才会当做时间数据进行处理
使用ts()函数可以把向量转化为时间序列数据:

常见错误
定义向量不要忘记加c,定义多个数据时记得要使用c()
使用函数要记得加括号,尽量多使用自动补齐功能
两个等号才是比较是否相等
获取数据
R获取数据一共有三种途径:
利用键盘来输入数据
通过读取存储在外部文件上的数据
通过访问数据库系统来获取数据
读入文件(一)
R读取文件
纯文本文件(.csv/.txt):
通常使用逗号,也可以使用空白分割
Read.table()函数可以读取一个纯文本文件,read.table(file=要读入的文件的名称;sep=指定文件使用的分隔符,默认是空白分割;header=代表在读取数据之后是否将数据的第一行作为变量的名称,而不是当成具体的值来处理,如果是,header=T,不是,header=F;skip=表示读取参数时,跳过部分内容,比如说可以跳过一些介绍性文字;nrows=用于读取文件的行数;na.strings=用于处理缺失值的信息)
x <- read.table(file = "input.txt")#需要在工作目录下才可以直接输入名称
如果不在工作目录下,可以利用setwd()函数来更改R的工作目录,或者使用文件的全路径:

可以使用head()和tail()函数读取文件的前几行或者是后几行数据,可以通过函数中选项参数n的数值来确定显示的行数

使用sep可以指定对数据进行分割,使数据格式变得整洁:

如果提前知道文件格式,也可以直接用read.csv()进行直接读取:

例如需要读取文件前100行的数据:


在此基础上使用skip和nrows两个参数相结合,就可以读取任意部分的数据,比如读取上述文件的第10-50行数据:
read.table("input 1.txt",header = T,skip=10,nrows = 50)
R在读取数据时,字符串数据会被默认读取成因子型数据。
读入文件(二)
如果一个纯文本文件并不在本机上,R可以支持读取网络文件,可以通过一些协议进行读取,只要将read.table()函数的选项参数file=文件的网络链接即可。R会将文件下载到本地。
如何读取非文本文件?
我们可以使用XML包进行读取,里面包含一个readHTMLTable()函数,可以用于读取网页中的数据。
R中的foreign包可以帮助导入其他软件的数据
或者可以直接导入剪切板的内容,直接将read.table()函数选项参数改为clipboard即可:

R可以直接读取压缩文件,并不需要解压缩:

如何写入文件
使用write.table()函数可以将数据写入文件,函数中,write.table(x=写入的数据,file=数据的存储路径及格式”)
如:
x=read.table("input.txt")
write.tale(x,file="H:/Rdata/newfile.txt")
也可以使用sep参数确定分隔符
write.table(x,file = "H:/RData/newfile.csv",sep=",")
这样就等同于做了一次数据转换,将文本数据转变成了表格数据
每一次加载数据,R会自动给每一行数据添加行号,为避免多次打开数据导致行号重复,可使用row.names参数
write.table(x,file = "H:/RData/newfile.csv",sep=",",row.names = F)
R会覆盖同名文件,使用append参数可以添加同名参数
R可以直接写入压缩文件:
write.table(mtcars,gzfile("newfile.txt.gz"))
读写excel文件
可以使用两种方式:
将excel文件另存为csv文件,再使用read.csv()打开
如:
read.csv("H:/RData/mtcars.csv",header = T)
或者将数据复制到剪贴板,使用readclipboard()函数将数据导入R中,在R中打开:
readClipboard()
read.table("clipboard",sep = '\t',header = T)
在R中也提供了很多包用于直接读写excel数据
比如:xlconnect包(这个包依赖于java,需要有java的环境),如果该R包无法安装,可以使用openxlsx包进行读取,如下:
library(openxlsx)
read.xlsx("H:/RData/data.xlsx",sheet = 1)
还可以使用openxlsx包的函数创建并写入新的excel文件,代码如下:
wb <- createWorkbook()
addWorksheet(wb,sheetName = 1)
x <- mtcars
writeData(wb,sheet = 1,x,startCol = 1,startRow = 1)
write.xlsx(x,"cars.xlsx")
这样就创建了具有mtcars数据的excel文件。
读写R格式文件
R提供了两种存储的方式,一种时候.Rds文件,一种是Rdata文件。
Rdata文件类似于工程文件,会存储所有导入的数据集和处理的数据
Rdse文件是保存数据集的文件,比如iris数据
使用saveRds命令可以将数据集保存为Rds格式
将readRds赋值给一个变量x,可以完成对Rds文件的读取。
使用load()函数可以直接打开Rdata文件。
数据转换(一)
首先,使用openxlsx包中的read.xlsx()函数打开mtcars.xlsx文件
read.xlsx('mtcars.xlsx',sheet = 1,startRow = 1)
将其赋值为car32变量
car32 <- read.xlsx('mtcars.xlsx',sheet = 1,startRow = 1)
可以使用is.data.frame()判断是否为数据框
is.data.frame(car32),结果是ture,说明数据是数据框
is.data.frame()函数还可以将数据转换为数据框格式
unlist()可以用于转化成列表
as.factor、as.vector可以用来转化成因子和向量。
数据转换(二)
如何对数据取子集?
可以使用索引的方式:
who <- read.csv("WHO.csv",header = T)
取该数据集的前50行,10列
who1 <- who[c(1:50),c(1:10)]
也可以不连续的提取,取该数据集的1,3,5,8行,2,14,18列:
who2 <- who[c(1,3,5,8),c(2,14,18)]
还可以使用逻辑值来进行筛选,比如使用which函数取出who数据集中continent列的值等于7的数据集合:
who3 <- who[which(who$Continent==7),]
还可以使用逻辑值的设置范围进行取值:
who4 <- who[which(who$CountryID>50 &who$CountryID<=100),]
取出who数据集中CountryID列的值在50到100之间的数据集合
可以直接使用subset()函数进行子集的提取
who4 <- subset(who,who$CountryID>50 &who$CountryID<=100)
在R中可以使用sample函数进行随机抽样
x <- 1:100
(设置一个x样本,数据范围在1到100)
sample(x,30)
(随机取x中的30个样本)
sample(x,30,replace = T)
(随机取x中的30个样本,但是是有放回的抽样,也就是说样本中可以有重复数字出现)
sample函数用于数据框时,如下
who[sample(who$CountryID,30,replace =F),]
随机取出了一个子集
如何删除固定行?最简单的就是使用负索引的方式,如下
mtcars[-1:-5,] 删除对应的列,逗号在前,删除对应的行
将列的值赋值给NULL,相当于清空该列的值
mtcars$mpg <- NULL
数据框如何进行添加与合并?
最简单的方法是使用data.frame()直接生成一个新的数据框
data.frame(USArrests,state.division)
如果单纯的想添加一列,可以用cbind函数
cbind(USArrests,state.division)
直接在数据后面添加一列state.division的数据
但是添加行不容易,因为使用Rbind()必须两者的列名是一样的,下面进行一个示范:
data1 <- head(USArrests,20) 取出前20行数据
data2 <- tail(USArrests,20) 取出后20行数据
rbind(data1,data2) 将两个行合并
在使用cbind和rbind的时候,数据必须有相同的行数和列数
如果数据集中有重复的数据应该如何处理呢?
假设取一个50个数据量的数据集的两个子集,容量分别为30,重复的数据有10
data1 <- head(USArrests,30)
data2 <- tail(USArrests,30)
使用rbind合并
data4 <- rbind(data1,data2)
使用duplicated(data4)判断哪些是重复值,取出重复值:
data4[duplicated(data4),]
加感叹号取出非重复的部分 (感叹号是取反的意思)
data4[!duplicated(data4),]
可以使用unique()函数一步完成非重复部分的提取
unique(data4)
数据转换(三)
数据框的翻转
使用t()函数可以实现数据框的转置,行转成列。列转成行
sractm <- t(mtcars)
翻转单独一行
可以使用rev()函数
rev(row.names(women))
women[rev(row.names(women)),]
先试用rev()函数翻转利用row.names()函数取得的行名,然后再将翻转后的行名作为索引,取出该行就行
如何修改数据框的值
比如将women数据框中的height列数据的值的单位由英寸转化成厘米
women$height*2.54
data.frame(women$height*2.54,women$weight)
先取出这一列,让数值全部乘以换算值,之后使用data.frame()函数重新组合成一个数据框
但这种方法并不高效
可以使用transform()函数
transform(women,height=height*2.54)
如果不想更改原来的数据,也可以直接用transform增加一列
transform(women,cm=height*2.54)

如何对数据框进行排序
Sort()对向量进行排序,返回的是排序后的结果向量,rev(sort())则是按照相反的顺序进行排序
Order()也可以对向量进行排序,但返回的是向量所在的位置,也即是向量中 元素的索引
如果想按照某一行的元素的大小顺序进行排序,利用order的话可以这样写:
order(mtcars$cyl)
mtcars[order(mtcars$cyl),]
如果想取相反的顺序,直接在数据前添加一个负号即可
mtcars[order(-mtcars$cyl),]
还可以进行多重条件下的排序
mtcars[order(mtcars$mpg,mtcars$disp),]
数据转换(四)
如何对数据框进行数学计算?
有很多种方法,以wordphones数据为例:
rs <- rowSums(WorldPhones)
cm <- colMeans(WorldPhones)
rowsums()和colmeans()函数可以计算行和还有列的平均数
使用cbind()函数可以直接在数据后面添加计算后的一列数据
total <- cbind(WorldPhones,total=rs)
使用rbind()函数可以直接在数据后面添加计算后的一行数据
total2 <- rbind(WorldPhones,meanvalue=cm)
在R中提供了apply系列函数
首先是apply()函数的用法,对数据框的每一行进行求和
apply(WorldPhones,MARGIN = 1,FUN =sum)
其中,wordphones代表要进行求和的数据集,margin是数据的维数,margin=1代表按行,margin=2代表按列,FUN参数代表使用的函数
对数据框的每一列进行求平均值
apply(WorldPhones,MARGIN = 2,FUN =mean)
对数据框的每一列进行求log值(对数值)
apply(WorldPhones,MARGIN = 2,FUN =log)
lapply()函数用法与apply()函数类似,不过返回值是列表,同理,sapply()也是返回值不同,sapply()函数返回的是向量或者矩阵。
以state.center数据为例,该数据是列表数据:
统计一下列表中元素的个数:
lapply(state.center,FUN = length)
因为列表中不存在行和列,所以不需要有margin参数,返回的结果值是一个列表
tapply()函数用于处理因子数据,根据因子来分组,然后根据分组来处理
以state.name和state.division为例,state.division作为因子数据进行分组:
计算每个类型区中州的数量
tapply(state.name,state.division,FUN = length)

接下来是数据的中心化与标准化处理(为了消除数据量纲对数据的影响),以state.x77数据为例:
数据的中心化,是指数据集中的各项数据减去数据集的均值
数据的标准化,是指在中心化之后再除以数据集的标准差,即数据集汇总的各项数据减去数据集的均值再除以数据集的标准差
在R中可以直接使用scale()函数进行中心化和标准化的处理,当scale()函数中的两个参数都为True时,就是做中心化(center=T)和标准化处理(scale=T)
scale(state.x77,center = T,scale = T)
经过中心化和标准化处理过后的数据,在绘制heatmap时会比较精确,对比性比较强。
数据转换(五)——reshape2包的使用(详细可参考R语言实战)
假设有x,y两个数据框数据如下:
x <- data.frame(k1=c(NA,NA,3,4,5),k2=c(1,NA,NA,4,5),data=1:5)
y <- data.frame(k1=c(NA,2,NA,4,5),k2=c(NA,NA,3,4,5),data=1:5)
使用merge()函数可以根据一个或多个共有的变量来进行合并,合并两组数据中共有变量列中相同的行
以k1作为共有变量合并:
merge(x,y,by = "k1")
以k2作为共有变量合并,并排除有NA的情况:
merge(x,y,by = "k2",incomparables = NA)
reshape2包是一个重构和整合数据的万能工具,可以把数据转化成任何想要的形式
使用airquality数据为例:
首先,为了输入方便,使用toloower()函数将列名改写成小写
names(airquality) <- tolower(names(airquality))
然后可以使用melt()函数融合数据,融合之后,每一行都是唯一的标识符~变量的组合:
aql <- melt(airquality)
也即是将宽数据变成长数据的过程
Id参数是用于告诉melt()函数哪一行或者那一列用作观测,而剩余的数据作为观测值
aql <- melt(airquality,id.vars = c('month','day'))
数据的重构使用dcast()或者acast()函数,处理数据框使用dcast()函数,acast()函数处理向量、矩阵或者数组:
Formula参数用于描述数据的形状,“~”在R中代表两者相关联,用以下代码即可重构数据
aqw <- dcast(aql,month day ~variable)
也可以只重构月数据,并对日数据求平均值,求一个月的平均值,还可以使用na.rm()去除缺失值:
aqw <- dcast(aql,formula = month ~variable,fun.aggregate = mean,na.rm=TRUE)
数据转换(六)——tidyr包的使用
Tidyr数据是一种很简洁的数据,数据的每一列代表一个变量,每一行代表一个观测,每一个观测的值在表中为一个单元格,也就是一个观测一个变量确定唯一的一个值。
接下来使用mtcars的数据来介绍一下tidyr包的函数用法
Mtcars数据是很典型的一个tidyr数据,先取数据的10行和3列,赋值到一个新的变量上。
tdata <- mtcars[1:10,1:3]
取数据的行名生成新的一个数据框(方便处理)
tdata <- data.frame(names=rownames(tdata),tdata)
gather函数:
gather(data,key,value…..),data参数是要处理的数据,key参数是宽数据变为长数据时,存放需要编码的变量的变量名称,需要自己定义,value参数是需要数据转换的变量的数值,也需要自己定义。
gather(tdata,kkey = 'key',value = 'value',cyl,disp,mpg)
处理前数据:
names mpg cyl disp
Mazda RX4 Mazda RX4 21.0 6 160.0
Mazda RX4 Wag Mazda RX4 Wag 21.0 6 160.0
Datsun 710 Datsun 710 22.8 4 108.0
Hornet 4 Drive Hornet 4 Drive 21.4 6 258.0
Hornet Sportabout Hornet Sportabout 18.7 8 360.0
Valiant Valiant 18.1 6 225.0
Duster 360 Duster 360 14.3 8 360.0
Merc 240D Merc 240D 24.4 4 146.7
Merc 230 Merc 230 22.8 4 140.8
Merc 280 Merc 280 19.2 6 167.6
处理后数据:
names key value
1 Mazda RX4 cyl 6.0
2 Mazda RX4 Wag cyl 6.0
3 Datsun 710 cyl 4.0
4 Hornet 4 Drive cyl 6.0
5 Hornet Sportabout cyl 8.0
6 Valiant cyl 6.0
7 Duster 360 cyl 8.0
8 Merc 240D cyl 4.0
9 Merc 230 cyl 4.0
10 Merc 280 cyl 6.0
11 Mazda RX4 disp 160.0
可以使用冒号,制定多列聚到同一列中,此处是将cyl与mpg先合并,然后在和disp组成一个数据框
gather(tdata,key = 'key',value = 'value',cyl:mpg,disp)
如果该在列名称前添加一个负号,代表不需要转换该列

也可以直接使用列的索引编号进行数据的重新构造
gather(tdata,key = 'key',value = 'value',2:4)

Spread函数则是将合并数据转化成tidyr形式的数据
例如,对之前合并的tdata函数进行分开:
gdata <- gather(tdata,key='key',value = 'value',2:4)
spread(gdata,key = 'key',value="value")
就可以还原数据:

Separate函数可以把一列拆成多个列:
例如我们创建一个数据框df
df <- data.frame(x=c(NA,"a.b","a.d","b.c"))
将数据按照“.”分成两列:
separate(df,col = x,into = c('A','B'))
对x这列进行分割,分割成A、B两列,默认识别分隔符
分割之前:

分割之后:

当出现多个分隔符而识别出现错误时,例如以下情况:
df <- data.frame(x=c(NA,"a.b-c","a-d","b-c"))
则可以利用sep函数指定分隔符,这样就能顺利分列:
separate(df,col = x,into = c('A','B'),sep="-")
可以使用unite函数将分开的列连接起来
unite(x,col ="AB",A,B,sep = "-")
x是需要处理的数据,col是连接后字段的名字,A,B是需要连接的字段,sep是用于连接的连接符
连接之后:

数据处理——dplyr包的使用
使用iris数据对dplyr包的用法做介绍,首先是filter函数
可以使用filter函数用指定条件对数据进行筛选,比如以下代码可以筛选掉花萼长度在7以下的数据,保留在7以上的数据
dplyr::filter(iris,Sepal.Length>7)
distinct函数可以用于去除重复行,相当于unique函数的功能
比如,可以使用rbind函数合并iris数据集的1-10行数据与1-15行数据,再使用第三条函数去除多余行:
dplyr::distinct(rbind(iris[1:10,],iris[1:15,]))
以上代码的运行结果只会保留,10-15行的数据,因为1-10行数据是重复的
Slice函数可以用于切片,可以取出数据的任意行:
dplyr::slice(iris,10:15)
以上代码可以取出iris数据的10-15行
Sample_n函数用于随机取样,例如:
dplyr::sample_n(iris,10)
代表在iris这个数据中随机抽取10行
Sample_frac表示按比例随机选取,比如抽取源数据的10%的数据,是指百分比:
dplyr::sample_frac(iris,0.1)
arrange函数用于排序,比如将iris数据按照花萼长度进行排序(正向),可写成:
dplyr::arrange(iris,Sepal.Length)
如果加上前面加上负号则是进行反向排序(从大到小)
dplyr::arrange(iris,-Sepal.Length)
关于dplyr的统计函数:
可以使用summarise()函数进行统计,比如统计花萼长度的平均值:
summarise(iris,avg=mean(Sepal.Length))
统计花萼长度的和:
summarise(iris,total=sum(Sepal.Length))
R中一个非常有用的负号:%>% 这个符号是链式操作符,它的功能是用于实现将一个函数的输出传递给下一个函数,作为下一个函数的输入,在键盘上可以用Ctrl shift M的快捷键打出来,有点类似于“且”的概念。
比如,先使用head函数取出数据集的前20行,再使用链式操作符,可以将这20行数据作为下一个命令的输入,如下一个命令是tail(10),也就是取出倒数10行,那么就会取出这20行数据的第11-20行:
head(mtcars,20)

head(mtcars,20) %>% tail(10)

Group_by函数可以对数据进行分组,例如根据species这列对数据进行分组:
如果用链式操作符,代码可以改为:
iris %>% group_by(Species)
还可以结合summarize统计函数进行进一步的计算,例如可以计算每一种类型鸢尾花品种的花萼宽度的平均值:
iris %>% group_by(Species) %>% summarise(avg=mean(Sepal.Width))
在此基础上,还可以使用链式操作符,对宽度的平均值进行排序:
iris %>% group_by(Species) %>% summarise(avg=mean(Sepal.Width)) %>% arrange(avg)
mutate函数可以添加新的变量
使用mutate函数在iris数据中增加一行,该行数据是花萼与花瓣长度的总和:
dplyr::mutate(iris,new=Sepal.Length Petal.Length)
下面介绍一下的dplyr包对于双表格的操作:
比如将两个表格整合到一起,先创建两个数据框,分别是a,b
a=data.frame(x1=c('A','B','C'),x2=c(1,2,3))
b=data.frame(x1=c('A','B','D'),x2=c(T,F,T))
首先是左连接,left_join(),以x1列为基础进行连接
dplyr::left_join(a,b,by="x1")

然后是右连接,right_join(),是以b的x1列为基础进行连接,

然后是内连接和全连接,内连接是取x1的交集,全连接是取x1的并集
内连接:
dplyr::inner_join(a,b,by="x1")

全连接:
dplyr::full_join(a,b,by="x1")

半连接相当于根据右侧表的内容对左侧表进行过滤,也就是将两个表的数据的交集取出来:
半连接:
dplyr::semi_join(a,b,by="x1")

反连接是输出两个表的补集部分,也就是取出数据框a中b不含有的行:
dplyr::anti_join(a,b,by="x1")

以上两个函数是互补的函数,一个是取共有的,一个是取没有的。
下面来看几个数据框的合并操作
首先定义数据first变量,变量取mtcars数据前20行,second变量取第10-30行,由于slice函数取出的数据不含行名,需要在取出数据前用mutate函数添加一列行名
mtcars <- mutate(mtcars,names=row.names(mtcars))
first <- slice(mtcars,1:20)
second <- slice(mtcars,10:30)
然后验证函数
Intersect()取交集:
> intersect(first,second)
Union_all()取并集:
dplyr::union_all(first,second)
Union()取非冗余的并集,去除掉重复部分再进行合并:
dplyr::union(first,second)
setdiff()取数据的补集,也就是取出frist数据中second数据没有的部分:
setdiff(first,second)
函数介绍——R语言中的函数

使用lm()函数进行回归分析,例如研究state.x77这个数据,研究犯罪率与其他指标的关系:
首先将数据转化为数据框,因为lm()函数只能对数据框进行操作
state <- as.data.frame(state.x77[,c('Murder','Population','Illiteracy','Income','Frost')])
再使用lm()函数进行回归分析,研究人口、文盲率、收入以及天气对犯罪率的影响:
fit <- lm(Murder ~ Population Illiteracy Income Frost,data=state)
使用summary()得出统计结果
summary(fit)

后面的*代表数据的显著性,数据中,文盲率的回归系数是4.14,表示其他数据不变时,文盲率上升1%,则犯罪率上升4.14%,且回归系数在p<0.001的情况下,显著不为0,也就是显著。而如果数据不显著,则说明两者没有直接关系。
R函数的返回值:
ls()返回当前环境中的对象,也就是有多少变量
sys.date()返回当前系统时间
rm()删除指定的变量,但是这个函数是直接删除,不会有返回
使用函数要注意输入数据的格式

可以使用help()函数查看每个函数的帮助文档
函数介绍——函数的选项参数
一般函数的选项参数可以分成三个部分:输入控制部分;输出控制部分;调节部分
输入控制部分负责告诉用户函数能接受哪种类型的数据,这个选项参数往往出现在函数的第一位,比如说,有些函数的第一位选项参数是“file”说明使用这个函数你需要输入一个文件;如果是“data”则是需要输入一个数据框;“x”代表单独的一个对象,一般都是向量,也可以是矩阵或者列表;“x和y”代表函数需要两个输入变量;“x、y、z”函数需要三个输入变量;“formula”输入的是公式;(具体查看每个函数的帮助文档)
输出控制部分
调节参数:

选项接受哪些参数:

函数介绍——数学统计函数
(1)概率函数
概率论是统计学的基础,R有许多用于处理概率,概率分布以及随机变量的函数,R对每一个概率分布都有一个简称,这个名称用于识别与分布相联系的函数,这部分涉及到很多统计学基础的理论知识,比如随机试验、样本空间、对立与互斥、随机事件与必然事件、概率密度、概率分布等。
R中的概率函数(正态分布):d前缀—概率密度函数;P前缀—概率分布函数;q前缀—分位数函数(分布函数的反函数);r前缀—产生相同分布的随机数
Norm表示正态分布

例如生成一组符合正态分布的随机数,这组数据均值为15,标准差为2,总共100个数据:
> rnorm(n=100,mean = 15,sd=2)
R中的概率函数(离散分布):同样的道理,在这些分布缩写前面加上d、p、q、r就变成函数

这些分布函数可以帮助我们在R中绘制各种分布函数图。
R中如何生成随机数:
最简单的是runif()函数,可以生成0-1之间的随机数
生成50个0-1之间的随机数
> runif(50)
如果想生成0-1之外的随机数,可以通过修改选项参数来更改
runif(50,min=1,max = 100)
这样就能生成1-100以内的随机数了
Set.seed()函数可以绑定随机数,当输入Set.seed()函数时,回到最初的随机数:

每个Set.seed()号码对应的随机数是相同的,这个功能主要是可以在研究发表时重现随机分组,以保证在不同设备上也能独立获得相同的结果。
函数介绍——描述性统计函数(R语言实战p.131)
首先是summary()函数,运行一次该函数,就可以对数据进行详细的统计。
先自定义一个变量,把mtcars的数据赋值给这个变量:
myvars <- mtcars[c("mpg",'hp','wt','am')]
使用summary()函数计算变量的数据,结果包括最小值,最大值,四分位数和数值型变量的均值。

在回归分析中也会大量用到summary()函数
Fivenum()函数和summary()函数类似,但可以返回5个基本的统计量,包括最小值,四分位数、中位数、上四位数、最大值。
fivenum(myvars$hp)
这里使用了$符取出单个数据列进行统计。
Hmisc包中的describe()函数也可以计算统计量,可以返回变量和观测的数量、缺失值和唯一值的数目、以及平均值、分位数、已经五个最大的值和五个最小的值
describe(myvars)
pastecs包中有一个stat.desc()函数可以计算种类繁多的描述性统计量,stat.desc()函数的选项参数中,x是一个数据框或者是时间序列
install.packages("pastecs")
library(pastecs)
stat.desc(myvars)

如果设置base选项等于true,那么就会计算一些基本值,包括全部值的数量、空值以及缺失值的数量、最小值、最大值、值域、还有总和
stat.desc(myvars,basic = T)
如果设置desc选项等于true,那么就会计算一些描述值,包括中位数、平均数、平均数的标准误、平均置信度为95%的置信空间、方差、标准差、以及变异系数等
> stat.desc(myvars,desc = T)
默认情况下,两个选项参数都是T
如果设置norm为T,那么就会计算一些统计值,包括正态分布统计量、偏度和峰度等
stat.desc(myvars,norm = T)
psych包中也有一个describe()函数,可以计算非缺失值的数量、平均数、标准差、中位数、截尾的均值、最大值、最小值、偏度和峰度等等内容
截尾的均值是去掉两头的数据取均值,就像打分时,去掉一个最低分、去掉一个最高分,然后中间数据求均值。
describe()函数可以通过设置trim参数,设置去除比例,如trim=0.1,则是去除数据中最高和最低的10%的部分
当两个包的函数名一样时,后面载入的包的函数会覆盖前面载入的包的函数,如果要使用前面一个包的函数,只需要在包后加冒号再使用即可,如:
Hmisc::describe()
Aggregate()函数可对数据进行分组描述,能够对数据按照指定的分组信息进行统计,将分组信息通过一个列表指定出来即可
例如我们使用mass这个包中的cars93数据集【93年许多不同汽车的指标】
> library(MASS)
以制造商这一列数据为例,
aggregate(Cars93[c("Min.Price","Max.Price","MPG.city")],by=list(Manufacturer=Cars93$Manufacturer),mean)
这样就是根据汽车制造商来对数据进行分组统计,计算每个汽车制造商产品平均的价格
还可以根据产地:
aggregate(Cars93[c("Min.Price","Max.Price","MPG.city")],by=list(origin=Cars93$Origin),mean)

还可以将mean函数替换成其他函数,比如sd,计算数据的标准差
aggregate(Cars93[c("Min.Price","Max.Price","MPG.city")],by=list(origin=Cars93$Origin),sd)
也可以一次性使用多个分组条件,只需要在列表中添加即可,例如同时使用产地和制造商来分组:
aggregate(Cars93[c("Min.Price","Max.Price","MPG.city")],by=list(origin=Cars93$Origin,Manufacturer=Cars93$Manufacturer),mean)
aggregate函数的缺点是一次只用使用一个统计函数,比如只能返回平均值、方差等,可以使用一些扩展包来进行分组计算并实现返回多种描述性统计量
首先是doBy包中的summaryBy()函数,summary_by(data, formula,id = NULL,FUN = mean..)
在波浪线左侧是需要分析的数值型变量,直接写数据框中的列的名字就可以,不需要添加引号;不同变量之间用 号表示;右侧的变量是类别型的分组变量;data参数指定数据集,fun参数指定统计函数,也可以是自定义函数:
summaryBy(mpg hp wt~am,data=myvars,FUN = mean)

Fun参数后面可以接多个统计函数,如
summaryBy(mpg hp wt~am,data=myvars,FUN = c(mean,sd,sum))

Psych包中的describe.by()函数和describe()函数能够计算相同的统计量,但是describe.by()函数可以通过分组来计算,只需要添加一个分组的列表,直接给定一个list即可:
describe.by(myvars,list(am=mtcars$am))

describe.by()适合详细查看每一个分组的统计值,但缺点是给出的统计值是固定不变的,没办法使用自定义的函数
函数介绍——频数统计函数
频数在数据分析中是非常重要的一个概念,因为经常需要进行分组统计,比较不同组之间的差异,这些都要涉及到频数的统计。因子是专门用来进行分组的,有因子才能分组,分组之后才能进行频数统计。
首先介绍一下R如何对数据进行分组:
如果一个数据本身就是因子,那么直接就可以进行分组,例如mtcars数据集,里面的“cyl”这一列数据直接就可以作为因子,依据气缸数的不同来进行分组。
用as.factor()函数将这列转换为因子数据:
cylfactors <- as.factor(mtcars$cyl)
然后可以使用split()函数对数据进行分组:
myvars <- dplyr::mutate(myvars,cylfactors1=cylfactors)
split(myvars,myvars$cylfactors1)
还可以使用cut()函数(如果没有明显的分类),cut可以对连续的数据进行切割,使用cut()函数对“mpg”这列进行分割,根据10-50切成10份,就是以步长为10,每十下一切:
cut(myvars$mpg,c(seq(10,50,10)))

此处seq()函数的选项参数是:seq(from=,to=,by=组距)
在分组之后,就可以用table()函数进行频数的统计,table()函数可以计算频数表。
table()函数的使用比较简单:
table(myvars$cylfactors1)

cut()函数的结果也可以使用table()来统计:
table(cut(myvars$mpg,c(seq(10,50,10))))

以上就是频数统计的做法
用频数除以总数就是频率值,R中可以直接使用prop.table()函数计算频率值:
prop.table(table(myvars$cylfactors1))

频率值*100就是百分比的结果。
那么二维的数据框如何进行频率统计呢?
可以使用table()或者是as.table()函数,选项参数输入两个因子就可以计算二维数据的频率了。
这里我们以vcd包中的风湿病数据集(arthritis)进行示范。
Arthritis数据集中的“treatment”、“sex”以及“improved”列都可以作为因子数据,因为它们是类型量,我们取两个作统计:
table(Arthritis$Treatment,Arthritis$Improved)
返回的结果是一个二维的列联表,横向是安慰剂组与治疗组的区分,每一列分别是“没有效果”、“有一些效果”、“很有效果”的区分:

如果变量太多,我们也可以先使用with()或者是attach()函数先加载数据:
with(data = Arthritis,table(Treatment,Improved))
这样就不用反复的书写变量名称
处理二维列联表还可以使用xtabs()函数,这个函数的好处是它的选项参数使用的是formula参数,这样就可以根据需要写成多种公式。
同样是“treatment”以及“improved”列,使用xtabs()函数计算如下:
xtabs(~ Treatment Improved,data = Arthritis)
formula参数是可以省略的,结果和with()函数的一致

对于二维列联表我们还可以使用margin.table()和prop.table()函数分别计算边际频数与比例(边际频率),边际频数的意思就是单独按照行或者列的数据进行处理。
我们将xtabs()的结果保存到变量x中,再使用margin.table()函数统计一下x:
x <- xtabs(~ Treatment Improved,data = Arthritis)
margin.table(x)
返回值只有一个,代表返回的是所有的结果,这里需要给定一个边际值,1或者2,1代表行,2代表列:
margin.table(x,1)

使用prop.table()函数计算比例(边际频率):
prop.table(x,1)

从边际和比例的计算结果可以看出,有治疗组有51%的比例是效果显著的,比边际频数41要大,说明药物是有用的。
再将margin.table()参数换成2,则是按列进行统计
margin.table(x,2)
addmargin()函数可以直接将边际的和添加到频数表中
addmargins(x)

这个函数也可单独计算行、列
addmargins(x,1)

addmargins(x,2)

我们也可以计算三维的列联表,加多一个参数就可以:
xtabs(~ Treatment Improved Sex,data = Arthritis)

结果看起来有点乱,这时候我们可以使用ftable()函数,它能将结果转换为一个平铺式的列联表
y <- xtabs(~ Treatment Improved Sex,data = Arthritis)
ftable(y)

函数介绍——独立性检验函数
独立性检验是根据频数信息判断两类因子彼此相关或相互独立的假设性检验,所谓独立性就是指变量之间是独立的,没有关系。
根据分组计算的频数表就可以进行独立性检验。
主要介绍三种检验方法:卡方检验、Fisher检验、cochan-mantel-haenszel检验

p-value就是probability的值,它是一个通过计算得到的概率值,也就是在原假设为真实,得到最大的或者抄书做得到的检验统计量值的概率。一般将p值定位到0.05,当p<0.05时,拒绝原假设(也就是假设成立),p>0.05是不拒绝原假设。
还是使用arthritis数据集,探究药物治疗有没有成效,检验“treatment”和“improved”是不是相互独立的,如果相互独立,说明二者没有关系,药物治疗没有作用,反之则是有效果。
先使用table()函数计算两者的频数:
mytable <- table(Arthritis$Treatment,Arthritis$Improved)
接下来就可以直接使用chisq.test()函数进行卡方独立检验,直接将结果输入这个函数就行:
chisq.test(mytable)
结果如下,p值约等于0.0014小于0.05,说明两者不是独立的,两者有关系,治疗是有效果的:

这种待检测的变量之间没有顺序的关系,调整两者的顺序,结果是一样的:
mytable <- table(Arthritis$Improved,Arthritis$Treatment)
chisq.test(mytable)

Fisher精确检验:
Fisher精确检验使用的函数是Fisher.test(),同样是进行独立性检验,但是与卡方检验不同的是,Fisher精确检验的原假设是:边界固定的列联表中行和列相互独立的。
还是同样的例子,采用Fisher精确检验:
mytable <- xtabs(~Treatment Improved,data = Arthritis)
> fisher.test(mytable)
结果为p值约等于0.0013,Fisher检验适合小样本的检验,精度低于卡方检验:

cochan-mantel-haenszel检验:
cochan-mantel-haenszel检验使用的函数是mantelhaen.test(),该鲜艳的原假设是两个名义变量在第三个变量的每一层中都是条件独立的。这个检验需要三个变量,此处我们来检测一下
“treatment”、“sex”以及“improved”之间的关系:
首先计算三个变量的列联表,使用xtabs()计算:
mytable <- xtabs(~Treatment Improved Sex,data = Arthritis)
mantelhaen.test(mytable)
结果为p值约等于0.0006,小于0.05,也就说明,药物治疗和改善情况在性别的每一个水平上不独立(因为这里使用了性别做第三层分类量):

如果调整变量顺序,反映的结果是有差别的:
mytable <- xtabs(~Treatment Sex Improved,data = Arthritis)
mantelhaen.test(mytable)


结果为p值约等于0.14,大于0.05,也就说明,药物治疗和性别在改善情况的每一个水平上独立(因为这里使用了改善情况做第三层分类量)
函数介绍——相关性分析函数
相关性分析是指对两个或者多个具备相关性的变量元素进行分析,从而衡量两个变量因素的相关密切程度。相关性元素之间需要存在一定的联系或者概率才可以进行相关性分析,简单来说就是变量之间是否有关系(也就是说需要先进行独立检测之后才能进行相关分析)。
相关系数的大小表示相关性的大小,相关系数包括:pearson相关系数、spearman相关系数、kendall相关系数、偏相关系数、多分格相关系数和多系列相关系数
与独立性检验不同,相关性分析中每种方法都没有独立的函数,这里面计算相关性系数都使用同一个函数:cor()函数。
cor()函数可以计算三种相关性系数,包括pearson相关系数、spearman相关系数和kendall相关系数,具体使用哪种方法可以使用选项参数中的参数method来指定(默认是用pearson相关系数),;函数中还有一个use选项,用于指定如何对待缺失值,是不处理还是删除等。
此处我们使用state.x77数据作为实例数据(这是一个矩阵数据):
cor(state.x77)
部分结果截图:

一般相关数据都是在[0,1]之间,数值越大越相关,正负号表示是正相关还是负相关
除了cor()函数之外,还有一个cov()函数可以用来计算协方差,携房产可以用来衡量两个变量的整体误差
例如我们定义两个变量:x,y
x <- state.x77[,c(1,2,3,6)]
y <- state.x77[,c(4,5)]
再使用cor()函数计算两者之间的相关系数:
cor(x,y)
结果看起来会比较整洁、清爽:

cor()函数只能计算三种相关系数,其他相关系数的计算可以通过R的拓展包来实现:
可以使用“ggm”这个包中的pcor()函数计算偏相关系数。
偏相关系数是指在控制一个或者多个变量时,剩余其他变量之间的相互关系:
pcor()函数需要输入两个重要的参数,第一个参数是一个数值向量(前两个数值表示要计算相关系数的下标,其余的数值为条件变量的下标),第二个参数是cov()函数计算出来的协方差结果
比如此处我们想控制收入水平、文盲率和高中毕业率的影响,看人口(第一列)和谋杀率(第五列)之间的关系(先获取列名,不然容易混):
colnames(state.x77)

pcor(c(1,5,2,3,6),cov(state.x77))
偏相关系数的结果:
[1] 0.3462724
函数介绍——相关性检验函数
在进行相关性分析之后,对相关性进行检验,cor.test()函数可用于相关性的检验,该函数有四个比较重要的选项参数,其中x和y是需要检测的相关性变量,alternative是用来指定进行双侧检验还是单侧检验,two.sided代表分别检测正负相关性,greater代表正相关,less代表负相关。Method选项用于指定用哪种相关系数,可选的有pearson相关系数、spearman相关系数、kendall相关系数。
我们使用state.x77数据检测一下谋杀率与文盲率之间的关系
cor.test(state.x77[,3],state.x77[,5])

置信区间:confidential interval,是指样本统计量所构造的总体参数的估计区间,在统计学中,一个概率样本的置信区间是对这个样本的某个总体参数的区间估计,置信区间展现的是这个参数的真实值有一定的概率落在测量结果周围的程度,置信区间给出的是被测量参数的测量值的可信程度(也就是说,光给出概率还不行,还要给出概率发生的范围)
Cor.test()只能一次性检测一组变量的关系,psych包中的corr.test()函数可以一次性进行多个变量的检验,这个函数还可以进行递归操作
corr.test(state.x77)
函数不仅计算了相关系数,还隔出了检测值:

如果想进行偏相关系数的检验,可以使用ggm包中的pcor.test()函数,先计算偏相关系数
pcor(c(1,5,2,3,6),cov(state.x77))
其中,x是pcor()函数计算的偏相关系数,然后是要控制的变量数,最后就是样本数
x <- pcor(c(1,5,2,3,6),cov(state.x77))
pcor.test(x,3,50)
返回三个值,分别是t检验,自由度和p value

分组数据的相关性检验,这种分组的检验可以使用t检验,t检验使用t分布理论,推论差异分布的概率,从而比较两个平均数的差异是否显著。主要用于样本含量较小,一般小于30个,总体标准差未知的正态分布数据
这里我们使用MASS包中的UScrime数据集,它包含了1960年美国47各州的刑罚制度对犯罪率的影响。
首先使用t.test()进行独立样本的t检验,t.test(y~x),y是一个数值型变量,x是类别型变量,
t.test(Prob ~ So,data = UScrime)

P<0.05,因此可以拒绝南方各州北方各州拥有相同犯罪率的假设。
如果不满足正态分布,就需要用非参数的方法,非参数检验在总体方差未知或者知道甚少的情况下,利用样本数据对总体分布形态等进行推断的方法,由于非参数检验方法在推断过程中不涉及有关总体分布的参数,因而得名为“非参数检验”
参数检验是在总体分布形式已知的情况下,对总体分布的参数如均值、方差等进行推断的方法,也就是数据分布已知,比如满足正态分布
函数介绍——绘图函数
R语言四大作图系统
基础绘图系统、lattice包、ggplot2包、grid包
可以使用demo(graphics)命令显示出R内置绘图函数可以做的一些图
R基础绘图系统包括两种绘图,高级绘图是一步到位可以直接绘制出图,而第几回吐,不能单独使用,必须在高级绘图产生图形的基础上,对图形进行调整,比如加一条线,加上标题文字等。
对于绘图函数要注意的是输入数据的格式
Plot()函数
Plot()函数可以接受一个单独的数值向量,例如:
plot(women$height)
绘制的是散点图
如果输入的数据是因子,绘制出来的则是直方图:
plot(as.factor(women$height))

再使用mtcars数据集看看:
plot(mtcars$cyl)
直接绘图就是散点图:

转换为因子数据输入就是直方图:
plot(as.factor(mtcars$cyl))

如果输入数据是一个因子数据和一个数值数据,则是箱线图:
plot(as.factor(mtcars$cyl),mtcars$carb)

如果输入第一个是数值数据,第二个是因子,输出的是散列图:
plot(mtcars$carb,as.factor(mtcars$cyl))

如果两个输入数据都是因子,输出的就是棘状图:
> plot(as.factor(mtcars$cyl),as.factor(mtcars$carb))

Plot()函数还可以接受函数公式:
plot(women$height~women$weight)
输出的是二者的关系图:

还可以用plot()函数直接绘制线性回归的结果:
fit <- lm(height~weight,data=women)
fit的值是线性回归的结果:

直接用:
plot(fit)

Plot()函数支持多种属性的数据格式。
Par()函数:
Par()并不能直接接用来绘图,而是对绘图参数进行调整
par()
直接敲par(),会跳出关于绘图的所有参数设置
例如我们绘制mtcars数据的因子数据图时,加上颜色:
plot(as.factor(mtcars$cyl),col=c("red","green","blue"))
函数介绍——自定义函数
编写函数就是为了减少重复代码的书写,从而让脚本更加简洁高效,增加可读性
在R中,如果直接写函数不加括号,就会显示函数的源代码
一个完整的R函数需要包括函数名称、函数声明、函数参数、函数体
函数名称:函数的命名最好和函数的功能相关,可以使字母和数字的组合,但必须是字母开头
函数声明:利用function函数来声明,用来告诉R这个东西是函数

下面我们来编写一个简单的R函数,这个函数的功能是计算偏度和峰度值:
偏度(skewness),是统计数据分布偏斜方向和程度的度量,是统计数据分布非对称程度的数字特征
峰度(peakness; kurtosis),又称峰态系数,表征概率密度分布曲线在平均值处峰值高低的特征数

第一个选项参数x就是要计算的数据,是一个数值向量,第二个选项是na.omit,用于删除缺失值,默认取值为False,然后是大括号,用于写函数的主体
然后是逻辑判断:
如果有缺失值,那么x只取不包含缺失值的x的值,!是取反的意思,is.na()是取数据集中的缺失值。
然后我们定义m为数值向量x的平均值,n为数值向量x的长度,s为数值向量x的标准差
然后写下计算skew(偏度)的公式:
skew <- sum((x-m^3/s^3))/n
计算峰度的公式:
kurt <- sum((x-m^3/s^4))/n-3
最后使用一个return函数返回函数的值
return(c(n=m,mean=n,stdev=s,skew=skew,kurtosis=kurt))}
这里我们要输出的是向量的个数n,平均值m,标准差s,偏度值和峰度值
然后可以使用一下这个函数:

下面来介绍一下R中的循环控制函数:
函数内部通过循环实现向量化操作,循环的三部分:条件判断,是真是假,用于循环执行的结构,表达式
首先看一下for循环:
for(i in 1:10){print("hello,world")}
然后是while循环:
i=1;while(i<=10){print("hello,world");i=i 1;}
分号;表示一个语句完结
i=1;while(i<=10){print("hello,world");i=i 2;}
i=i 2时语句会少一半
if else的结构
score=70;if (score>60){print("passed")} else {print("failed")}
还可以简写ifelse
ifelse(score>60,print("passed"),print("failed")
这节课可以参考用来写d-dematel 函数
项目实操——数据分析实战
通过实际案例进行数据分析,了解数据分析的实质
项目实操——线性回归(一)
回归,通常指用一个或多个预测变量,也成自变量或者解释变量,来预测响应变量,也称因变量、标效变量或者结果变量的方法
回归分析主要用于分析自变量对因变量的影响
重点是:如何建立模型、抽象出数学公式、哪些因素与模型有关、需要利用多少样品、模型的准确率有多高、在实际运用中还是否有效?

最简单的线性回归:普通最小二乘回归法(OLS)

我们可以使用lm()函数来进行线性回归分析,lm是linear model,线性回归模型的简称
这个函数的格式是:
lm(formula, data, subset, weights, na.action, method = "qr", model = TRUE, x = FALSE, y = FALSE, qr = TRUE, singular.ok = TRUE, contrasts = NULL, offset, ...)
formula:是要进行拟合的模型形式,写成一个公式,例如,y ~ ax b
data:是要使用的数据集,是数据框的格式

一般在回归分析中,都喜欢用fit这个变量名来定义结果,寻找回归模型的过程被称为拟合
如果后面data参数中指定了数据集,那么前面公式中的变量就可以直接写变量名字(注意,因变量在波浪线左边,自变量在右边)
fit <- lm(weight ~ height,data = women)
回归结果,可以使用summary()函数查看详细的分析结果:

summary(fit)

首先是call这一列,是列出使用的回归的公式。
然后是residuals,表示残差,残差是真实值和预测值之间的差,例如数据第一行,真实的值是58,将58代入预测公式,得出的预测值y,y与58之间的差值就是残差,残差给出了四个值,最小值、最大值、中位值、四分之一的值、四分之三的值,这四个值越小,说明预测模型越精确。
Coefficients:系数项,intercept:截距项(当x等于0时,与y轴的相交点)
Estimate是项系数的值,pr就是pvalue,是假设x与y不相关时候的概率,这个值也是小于0.05比较好,residual standard error残差标准误,表示残差的标准误差,这个也是越小越好。
Multiple R-squared: 0.991, Adjusted R-squared: 0.9903
这两个值称为R方判定系数,是衡量模型拟合质量的指标,它是表示回归模型所能解释的响应变量的方差比例,比如此处,就代表这个模型可以解释99.1%的数据,只有0.9%的数据不符合这个模型,取值在0-1之间,值越大于好。
最后是F-statistic(F统计量),这个值说明模型是否显著,也是用pvalue来衡量,也是值越小越好。
得出回归模型是:
Weight=3.45*height-87.52
是一个一元一次方程
在线性回归的结果中,一般先看F统计量,如果F统计量不显著(pvalue不小于0.05),那么这个模型就没有价值了,需要重新进行拟合,如果小于0.05,再看R方差,模型能解释多少变量。
项目实操——线性回归(二)
线性拟合常用函数:

使用predict()函数可以用拟合模型对新的数据集进行预测
直接使用plot()函数可以对拟合结果进行绘图:
plot(fit)
会生成四幅图:残差拟合图、正态分布qq图、大小位置图以及残差影响图
Abline()函数可以绘制出拟合曲线,但这个命令属于低级绘图命令,必须在高级绘图的基础上完成,我们先绘制身高与体重的散点图:
plot(women$height,women$weight)
> abline(fit)
最小二乘法的原理就是找到一条直线(拟合直线),使残差平方和最小
一般拟合曲线很少是直线,大部分都是曲线,也就是多项式的回归
还是women这个数据集,我们用多项式回归试一下:
先定义一个fit2变量
将体重作为因变量,身高与身高的平方作为自变量
fit2 <- lm(weight ~ height I(height^2),data = women)
可以对比两次回归的曲线差异
plot(women$height,women$weight)
abline(fit)
这次使用lines()函数,这个函数能把点连成线,横坐标是身高数据,纵坐标是根据拟合模型的得出的预测值,为了增加比较的差异性,我们给第二个曲线增加颜色:
lines(women$height,fitted(fit2),col="red")

对比很明显,带有二次项的回归模型能够更好的拟合数据,使得更多的点落在曲线上
那么,三次项的回归模型效果是不是更好呢?我们再来拟合一下
fit3 <- lm(weight ~ height I(height^2) I(height^3),data=women)
plot(women$height,women$weight)
abline(fit)
> lines(women$height,fitted(fit2),col="red")
> lines(women$height,fitted(fit3),col="blue")
拟合结果如图:

结果表明,继续增加多项式可以提高拟合度,但是其实没有必要,因为用于拟合的数据集,只是用于建模的数据集,不一定适合真实的数据,过多的拟合也是纸上谈兵。
项目实操——多元线性回归
当预测变量不止一个时,简单现象回归就变成了多元线性回归,相当于求解多元方程,而且和方程式求解不同的是,这些变量的权重还不一样,有些大 ,有些小,有些或者是没有多大影响,下面我们来看一个案例:
以state.x77这个数据集为例,求解出一个拟合模型,然后可以根据某些指标预测出犯罪率:
首先我们将state.x77这个矩阵数据转化成数据框,因为lm()函数输入数据必须是数据框的格式
这里为了简化问题,我们只取四个指标进行回归分析:population、illiteracy、income、frost
states <- as.data.frame(state.x77[,c("Murder",'Population','Illiteracy','Income',"Frost")])
还是使用一个lm()函数,定义一个fit变量,用于存储模型结果
fit <- lm(Murder ~ Population Illiteracy Income Frost,data = states)

使用summary()函数查看模型的详细过程结果
> summary(fit)

我们可以使用coef()函数单独查看各项的系数,根据系数项和截距值就能写出拟合方程
coef(fit)

options(digits = 4)
可以通过options()的digits参数设置显示的位数

下面我们来列举一个更复杂的例子,在很多研究中,变量会有交互项,也就是变量相互之间并不是独立的,例如mtcars数据集,汽车的重量与马力之间存在着交互,质量大会影响到马力。交互项就是解释变量之间存在相关性
我们来拟合一下mtcars数据集中,每加仑汽油行驶里程数(mpg)变量与马力(hp)以及车重(wt)之间的关系
fit <- lm(mpg ~ hp wt hp:wt,data = mtcars)
summary(fit)

从结果中可以看见,车重与马力的交互项是非常显著的,这就说明响应变量mpg与其中一个预测变量的关系依赖于另一个预测变量的水平,其实就是说,每加仑汽油行驶里程数域与汽车马力的关系需要依赖汽车的不同而不同。
对于多元线性回归存在的一个问题就是,如果存在多个变量,就需要考虑变量之间的相互影响和回归时的组合关系,也就是如何从众多可能的模型中选择最佳的模型呢?
比如是不是需要包括所有的变量,还是需要去掉一些对模型贡献不显著的变量,是否需要添加多项式或者是交互项来提高拟合度呢?
那么对于以上的问题,我们可以使用AIC()函数来比较模型。
AIC()函数是An Information Criterion的简称,称为赤池信息准则,这个准则考虑了模型统计拟合度以及用来拟合的参数数目
计算得到的AIC值越小越好,越小说明模型用较少的参数就可以获得足够的拟合度
例如上面拟合犯罪度的案例
Ctrl UP调出历史代码列表
对比一下两个fit的拟合度
fit1 <- lm(Murder ~ Population Illiteracy Income Frost,data = states)
fit2 <- lm(Murder ~ Population Illiteracy,data = states)
AIC(fit1,fit2)

结果显示,fit2的拟合度更好,但是如果变量数过多,那么组合起来的拟合模型数将是巨大的,再用AIC()两两比较就不太可行了
这个时候,对于变量的选择可以使用逐步回归法和全子集回归法。
逐步回归法中,模型会依次添加或者删除一个变量直到达到某个节点为止,这个节点就是继续添加或者删除变量,模型不再继续变化。如果每次是增加变量,那么就是向前逐步回归,如果每次是删除变量,那么就是向后逐步回归。
全子集回归法是取所有可能的模型,然后从中计算出最佳的模型,很显然全子集回归法要比逐步回归法要好,因为会检测到所有的模型,把可能的模型都纳入考虑之中,但是缺点就是如果变量数太多,会涉及到大量的计算,运算会比较慢。
这两种方法都需要通过R的扩展包来实现,MASS包中的stepAIC()函数可以进行逐步回归法的计算
> library(MASS)
> states <- as.data.frame(state.x77[,c("Murder",'Population','Illiteracy','Income',"Frost")])
> fit <- lm(Murder ~ Population Illiteracy Income Frost,data = states)
> stepAIC(fit,direction = "backward")

Leaps包中的regsubsets()函数可以进行全子集回归的计算
但是拟合效果最佳但没有实际意义的模型是没有用的,所以我们始终要对数据有所了解。
项目实操——回归诊断
找到回归模型之后,我们仍需要解决以下的问题:
这个模型是否是最佳的模型?
模型最大程度满足OLS模型的统计假设?
模型是否经得起更多数据的检验?
如果拟合出来的模型指标不好,该如何继续下去?
我们需要从多个维度对回归分析的结果进行诊断
除了利用summary()函数统计出来的各个指标进行检验,还可以用plot()函数进行绘图,可以生成评价拟合模型的四幅图
下面我们来举个例子:
我们还是使用women这个数据集:
fit <- lm(weight ~ height,data = women)
par(mfrow=c(2,2))
plot(fit)
#par()函数可以修改plot()函数的绘图参数,par()函数的mfrow选项参数可以定制图形的分布,默认是一张图上显示一幅图,可以使这个参数等于c这个向量,mfrow=c(2,2)表示横排显示两幅,纵排显示两幅,这样就可以将四幅图显示在一个画面内。
不是所有的数据都可以使用OLS模型进行拟合的,需要满足以下的前提条件:
正态性:对于固定的自变量值,因变量需要呈正态的分布
独立性:自变量之间相互独立
线性:因变量与自变量之间为线性相关
同方差性:因变量的方差不随自变量的水平不同而变化,也可以称作不变方差

第一幅图是残差与拟合的图,这幅图用来表示因变量与自变量是否呈线性关系,图中的点是残差值的分布,线为拟合曲线。如果图中是一个曲线的分布,说明可能存在二次项的分布
第二幅图是R中比较常见的qq图,QQ图用来描述正态性,如果数据呈正态分布,那么在QQ图中就是一条直线
第三幅图是位置与尺寸图,用来描述同方差性,如果满足不变方差的假设,那么图中水平线周围的点应该是随机分布的
第四幅图是残差与杠杆图,提供了对单个数据值的观测,从图中可以看到哪些点偏差较远,可以用来鉴别离群点、高杠杆点和强影响点,高杠杆点表示它是一个异常的预测变量的组合;强影响点表示这个值对模型参数的估计产生的影响较大,如果删除或者转换就可能违背客观数据的事实
但是这四幅图没办法说明数据是否具有独立性
只能从收集的数据中验证,要判断收集的数据是否独立,我们可以拟合函数再加一个二次项,再绘制一次图看一下。
fit2 <- lm(weight ~ height I(height^2),data = women)
> opar <- par(no.readonly = TRUE)
> par(mfrow=c(2,2))
> plot(fit2)
结果明显好一些:

抽样法验证:
1、数据集中有1000个样本,随机抽取500个数据进行回归分析;
2、模型建好之后,利用predict()函数,对剩余的500个样本进行预测,比较残差值
3、如果预测准确,说明模型可以,否则就需要调整模型。
项目实操——方差分析(一)
方差分析称为analysis of variance,简称ANOVA,也称为变异数分析,用于两个及两个以上样本均属差别的显著性检验,从广义上来讲,方差分析也属于回归分析的一种,只不过线性回归的因变量一般是连续型变量,而当自变量是因子时,研究关注的重点通常会从预测转向不同组之间差异的比较,这就是方差分析。
方差分析会大量用在科学研究中,例如实验设计时,进行分组比较,例如药物研究实验,处理组与对照组进行比较
方差分析也分为很多种,比如单因素方差分析,双因素方差分析、协方差分析、多元方差分析、多元协方差分析(R语言实战这本书中有详细介绍)
方差分析多采用aov()函数进行分析,aov()函数的用法与lm()函数类似。下表中列出了R中aov()函数的符号用法:

各种方差分析公式的写法:

变量的顺序很重要:

项目实操——方差分析(二)
单因素方差分析:
这里我们使用multcomp包中的cholesterol数据集进行演示,这个数据集是50个患者接受降低胆固醇治疗的五种疗法的数据,50个患者分为10人一组,每天服用20克药物
这个方案只有治疗方案这一个因子,所以被称为单因素方差分析
首先使用
library(multcomp)
attach(cholesterol)
这里使用attach()函数将数据集的列写入内存,这样便可以直接使用数据集的每一页,而不用每次都使用$来引用数据集,也就是表明以下的命令都在数据集下进行。
table(trt)
使用table()计算每一列不同因子的频数

接下来使用aggregate()函数进行分组统计数据的平均值:
aggregate(response,by=list(trt),FUN=mean)
进行方差分析:
fit <- aov(response ~ trt,data = cholesterol)
summary(fit)

方差分析的结果主要看F值和P值,F值越大说明组间差异越显著,这里就是五组之间平均值的差别,而P值只是用来衡量F值,P值越小说明F值越可靠,同样可以使用plot()函数绘制方差的结果:
par(mfrow=c(2,2))
> plot(fit)

下面我们再使用lm()函数做一下方差分析,比较一下二者之间的差别:
因为线性拟合要求预测变量是数值型,所以当lm()函数的预测变量是因子时,就会将一系列与因子水平相对应的数值型对照变量来代替因子,不过P值和F值的意义是不变的
fit.lm <- lm(response ~ trt,data = cholesterol)
summary(fit.lm)

下面介绍一个单因素协方差的例子,选取multcomp包中的litter数据集,其中weight是这个数据集的响应变量:
首先使用table()函数统计分组情况:
table(litter$dose)
然后分组统计一下平均数:
aggregate(weight,by=list(dose),FUN=mean)

使用方差分析检验一下组间是否有显著的差异
越基础性的效应越需要放在表达式前面,最好首先是协变量,然后是主效应,以此类推:
summary(fit1)

F值的检验表明,怀孕时间与幼崽出生的体重有关,控制怀孕时间,药物剂量与体重相关,证明药物是有影响的,而不是随机产生的
双因素方差分析案例:
使用内置的toothgrowth数据集:
首先使用xtabs()函数统计频率,结果是一个二维的列联表,再统计一下平均值
xtabs(~supp dose)

aggregate(len,by=list(supp,dose),FUN=mean)

分组统计平均数的结果显示,剂量越小,二者的差异越明显
使用factor()函数将dose这列的数据转化为因子数据,在进行方差分析:
两个变量的方差分析再加上两个变量还有交互,所以进行方差分析的时候,应该用*号连接两个自变量:
fit <- aov(len ~ supp*dose,data = ToothGrowth)

使用“HH”包中的interaction.plot()函数可以非常直观的对这个结果进行可视化,而且对任意数据因子涉及的主效应和交互效应都进行了展示:
interaction.plot(dose,supp,len,type="b",col=c('red','blue'),pch=c(16,18),main='interaction between Dose and Supplement Type')

接下来是多元协方差分析的例子:
这里以MASS包中的UScereal数据集进行展示:
前面的预备操作和上面的类似,先使用attach()将数据集写入内存,然后将数据中的非因子型数据转化为因子,再进行方差分析。
attach(UScereal)
shelf <- factor(shelf)
aggregate(cbind(calories,fat,sugars),by=list(shelf),FUN=mean)
#aggregate只能接受一个变量进行分组,所以这里使用cbind()将三个因变量组合成一个数据框
这里有三个因变量,一个自变量
fit <- manova(cbind(calories,fat,sugars)~ shelf)
summary(fit)

可以使用summary.aov()函数分别查看每个变量的结果
项目实操——功效分析
这节课程我们讨论一下,在数据分析的筹备阶段,我们应该选择多少样本,在一个分析中,如果样本数量过小,那么就算pvalue值非常小,非常显著,也是不可信的。
功效分析(power analysis)可以帮助在给定置信度的情况下,判断检测到给定效应值时所需的样本量,反过来,它也可以在给定置信度水平的情况下,计算在某样本量内能检测到给定效应值的概率。

功效分析的理论基础:

第一类错误:弃真,第二类错误:存伪


所以我们根据要检验的显著性水平、功效和效应值来推算所需要的样品数,R中利用pwr包来进行功效分析。
在pwr中包含了多种功效分析的函数,根据不同的假设检验选择不同的函数:
下面介绍线性回归功效分析的案例
F2=R2/1-R2,即模型解释度(模型方差平方和ssr)与平均数解释度(误差平方和sse)之比,F2效应值越大,样本越小;
V=n-u-1为误差自由度,与样本数和自变量个数相关,误差自由度越搞,说明样本越多,房差越大,F2效应值越小,即解释度越小。
U为自变量个数,与误差自由度正相关,即个数越多,所需的样本越多
Power功效,一般小于0.95,但差距不大,排除假阴性的水平之,power越大,v就越大
pwr.f2.test(u=3,sig.level = 0.05,power = 0.9,f2=0.0769)

结果表明,v=184.2426,也就是说假定显著性水平为0.05,在90%置信度的情况下,至少需要185个受试者才可以。
下面介绍方差分析功效分析的案例
假设现在两组样品做单因素方差分析,要达到0.9的功效,效应值为0.25,并选择0.05的显著性水平,那么每组需要多少样品量呢?可以使用pwr.anova.test()函数进行分析:
其中选项K是组的个数,n是各组的样本大小也就是我们要求的样本量,f是效应值,sig.level还是显著性水平,power为功效水平:
pwr.anova.test(k=2,f=0.25,sig.level = 0.05,power = 0.9)

最终求得n=85.03,所以每一组中至少要有86个样本
项目实操——广义线性模型
在现实生活中,大部分数据都是无规则分布的,要通过数据分析找到规律。
广义线性模型扩展了线性模型的框架,它包含了非正态因变量的分析,在R中可以通过glm()函数进行广义线性回归分析(具体可以参考R语言实战p283)
这里我们使用breslow数据集进行演示,这个数据集是遭受轻微或严重性癫痫病的病人的年龄和发病数进行的记录
响应变量是后八周内癫痫病发病数sumy,预测变量为治疗条件(trt),年龄(age)以及前八周的基础癫痫病发病数(base),之所以包含年龄(age)以及发病数(base),是因为他们对响应变量有潜在的影响,属于协变量,在解释这些协变量之后,才能知道药物治疗对癫痫病的影响
data(breslow.dat,package="robust")
summary(breslow.dat)
接下来使用glm()函数进行泊松回归:
attach(breslow.dat)
fit <- glm(sumY ~ Base Age Trt,data = breslow.dat,family = poisson(link = 'log'))
summary(fit)

在线性回归中使用的函数在广义线性模型中同样可以使用,我们可以使用coef()函数查-/看回归结果的系数项:
coef(fit)

由于泊松回归的系数是对数形式,我们可以再对其取指数:
exp(coef(fit))

结果表明,当年龄增加一岁时,平均数的癫痫发病数将乘以1.023,也就是癫痫发病数的期望值将乘以1.023,这就代表,年龄的增加与较高的癫痫发病率存在关联。
项目实操——logistic回归
当通过一系列连续型或类别型预测变量来预测而执行结果变量时,logistic回归时一个非常有用的工具。
这里我们以affair包中的数据为例来阐述logistics回归的过程:
先导入数据集,再使用summary()函数做一个简单的统计,之后使用table()函数统计一下出轨的频数,prop.table()函数统计一下频率:
data(Affairs,package="AER")
summary(Affairs)
table(Affairs$affairs)
> prop.table(table(Affairs$affairs))

性别的频数:
prop.table(table(Affairs$gender))

Logistic回归需要结果是二值型的,所以我们将其转为二值型的结果,定义一个ynaffairs(年度出轨次数):
Affairs$ynaffairs[Affairs$affairs>0] <- 1
> Affairs$ynaffairs[Affairs$affairs==0] <- 0
可以用过head()函数查看字段是否添加成功

接下来将这个变量转化为因子:
Affairs$ynaffairs <- factor(Affairs$ynaffairs,levels = c(0,1),labels = c("NO","Yes"))

现在ynaffairs就属于二值型的变量,可以进行logistic回归分析了(这一步去其实是把离散值转化为二项分布)
使用glm()函数进行logistic回归分析(这个数据集的变量比较多,先使用attach()函数将数据集写入内存,使用R的自动补齐功能):
fit <- glm(ynaffairs ~ gender age yearsmarried children religiousness education occupation rating,data = Affairs,family = binomial(link=logit))
summary(fit)

根据回归结果,将不显著的变量去除,重新拟合一次:
> fit1 <- glm(ynaffairs ~ age yearsmarried religiousness rating,data = Affairs,family = binomial(link=logit))

由于两个回归结果是嵌套关系,可以使用anova对两者进行方差分析,对于广义线性回归,可以使用卡方检验:
anova(fit,fit1,test = "Chisq")
卡方检验结果值并不显著

证明两次结果差别不大,证明两次拟合的结果一样好,说明性别、教育程度等不显著变量不会显著的提高方程的预测精度。
在logistic回归中,响应变量是y=1的对数优势比,回归系数的含义是,当其他预测变量不变时,一单位预测变量的变化可能引起的响应变量对数优势比的变化,对回归系数取指数,就能回归正常的数值。

可以使用predict()函数根据拟合模型的结果(如此处的fit1)对新数据进行验证,首先创建一个包含你感兴趣的预测变量值的虚拟数据集,testdata,这里为了方便,我们都取数据的平均值。
testdata <- data.frame(rating=c(1,2,3,4,5),age=mean(Affairs$age),yearsmarried=mean(Affairs$yearsmarried),religiousness=mean(Affairs$religiousness))
> head(testdata)

testdata$prob <- predict(fit1,newdata = testdata,type = "response")
testdata

下面我们再看看年龄的影响,重新生成一个测试数据:
testdata <- data.frame(rating=mean(Affairs$rating),age=seq(17,57,10),yearsmarried=mean(Affairs$yearsmarried),religiousness=mean(Affairs$religiousness))
#这里seq(17,57,10)的意思是,17岁到57岁,以10为间隔生成年龄数,相当于一个以10为差值的年龄等差数列。
testdata$prob <- predict(fit1,newdata = testdata,type = "response")
testdata

结果显示,当其他变量的增长,婚外情的概率将从0.31降到0.10,利用这种方法可以研究每一个预测变量对结果概率的影响
项目实操——主成分分析
主成分分析和因子分析都是用来探索和简化多变量复杂分析的方法。主成分分析,也简称为PCA,是一种数据降维技巧,它能将大量相关变量转化为一组很少的不相关变量,这些无关的变量成为主成分,主成分其实是对原始变量重新进行线性组合,将原先众多具有一定相关性的指标,重新组合为一组的新的相互独立的综合指标。

R中内置的printcomp()函数可以进行主成分分析,这里我们使用psych包进行分析
主成分分析与因子分析的步骤:
数据预处理、选择分析模型、判断要选择的主成分/因子数目、选择主成分/因子、旋转主成分/因子、解释结果、计算主成分或因子得分。
这里我们使用USjudgeratings数据集进行举例分析:
选择PCA分析,通过绘制碎石图选择需要的主成分数目:
fa.parallel(USJudgeRatings,fa="pc",n.iter = 100)

接下来使用principle()函数进行主成分分析,nfactors是主成分因子的数目,rotate是旋转角度,scores表示是否要计算主成分得分,默认为不需要:
pc <- principal(USJudgeRatings,nfactors = 1,rotate = "none",scores = FALSE)

这就是PCA分析的结果,其中,pc1栏是指观测变量与主成分的相关系数,如果nfactors=2或者3,那么还会有pc2、pc3等主成分,h2栏指成分公因子的方差,是主成分对每个变量的方差解释度,u2一栏是成分唯一性,方差不能被主成分解释的比例,proportion var表示每个主成分对数据集的解释程度,这里可以看到第一主成分pc1解释了所有变量84%的方差,我们将score参数设置为true,就可以获得每个变量的得分
接下来我们使用Harman23.cor数据集进行分析:

在这个数据集中,数据是由变量的相关系数组成而非原始的数据集,先进行筛选主成分因子,利用平行分析得出碎石图:
fa.parallel(Harman23.cor$cov,n.obs = 302,fa="pc",n.iter = 100)

只有两个x在y=1之上,所以选择两个主成分因子,接下来进行主成分的分析
下面介绍一下主成分的旋转(这里只展示代码,具体的统计学知识查看统计学书本以及R语言实战p303)
pc <- principal(Harman23.cor$cov,nfactors = 2,rotate = 'Varimax')
项目实操——因子分析
探索性因子分析法(exploratory factor analysis),简称EFA,是一系列用来发现一组变量的潜在结构的方法,它通过寻找一组更小的、潜在的或隐藏的结构来解释已观测到的、显式的变量间的关系。




下面列举一个因子分析的案例:
这里我们使用factanal()函数进行因子分析,使用ability.cov数据集进行演示:
首先对数据集进行处理,利用option()函数将数据保留两位小数
options(digits = 2)
covariances <- ability.cov$cov#定义covariences变量,取数据集中的cov列,因为ability.cov数据集是一个列表
转化为系数矩阵(原来表里面的数据是方差,然后用cov2cor()函数转化为相关系数):
correlations <- cov2cor(covariances)

同样使用fa.parallel()函数判断参与分析的因子个数,这里我们把选项参数fa设置为‘Both’表示即研究主成分也研究因子分析:
fa.parallel(correlations,fa="both",n.obs = 112,n.iter = 100)
n.obs是观测的数量,也就是样本数,可以通过abilitycov$n.cobs计算得出

观察图中结果,显示要提取两个因子。FA分析是看特征值数大于0,pc看特征值数大于1
fa <- fa(correlations,nfactors = 2,rotate = "none",fm="pa")
#nfactors是指因子数,rotate是指需不需要旋转,fm是用于进行因子分析的方法

正交旋转法:两个因子之间不相关,斜交旋转法:两个因子之间相关
(后面的多看书-R语言实战)
项目实操——购物篮分析
下面我们使用arules包以及groceries数据集进行演示:
由于这个数据没有办法直接在r中展示出来,我们需要使用inspect()函数查看数据集的内容,展现出来的效果是类似于商品小票一样的数据
使用arules包中的apriori()函数进行分析建模,这个函数就相当于线性回归分析中的lm()或者是aov()函数,是关联规则挖掘算法
Support=0.01表示最小支持度是0.01,confidence=0.5代表最小置信度是50%
fit <-apriori(Groceries,parameter=list(support=0.01,confidence=0.5))
通过summary()函数来查看统计信息,用inspect()函数查看给出的规则:

结果表明,在酸奶出现的地方,购物小票中就一定会出现纯牛奶,支持度达到了0.01,说明两者的关联很强。
,免责声明:本文仅代表文章作者的个人观点,与本站无关。其原创性、真实性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容文字的真实性、完整性和原创性本站不作任何保证或承诺,请读者仅作参考,并自行核实相关内容。文章投诉邮箱:anhduc.ph@yahoo.com






