sci如何作图(零基础用Pymol绘制蛋白质-小分子结合模式图)

文 / 利刃君
微信ID / ziyuanliren666
全文共2700字,推荐阅读时间7分钟。
本次教程以TRK-A蛋白与其原配体为例绘制蛋白质-小分子结合模式图。TRK-A晶体结构由PDB数据库(https://www.rcsb.org/)检索下载(PDB ID:6PL1),前期我们以Schrodinger软件进行了分子对接,其中:
①小分子配体与氨基酸Glu590、Met592、Asp668形成氢键作用;
②小分子配体与氨基酸Phe589、Phe669形成π-π堆积作用;
③小分子配体与氨基酸Phe669形成阳离子- π作用;
④小分子配体与氨基酸Met592、Tyr591等多个氨基酸形成疏水作用。
本次教程需要使用Pymol(需要center_of_mass.py插件)、Photoshop(PS)/PowerPoint(PPT)这三款软件。先来看一张成品图。

注:center_of_mass.py插件可由文末获取,或者从PyMolWiki网站自行下载。需要Pymol软件及安装教程可翻看利刃君历史消息获取。
-壹-
导入文件点击File-open…导入本次要处理的6PL1.pdb文件,也可以通过命令行的方法导入文件,利刃君这里通过命令行的方法导入文件,方便同时设置好工作目录。
在命令行输入以下命令(Pymol>表示这是Pymol的命令),输入完成后需要按回车执行命令。


然后点击Display-Sequence显示蛋白质序列及配体信息。此时,软件界面如下图所示。

-贰-
初步修饰我们可以更改蛋白的显示颜色,单击Viewer界面右侧菜单所示对象6pl1文本右侧的“C”,选择”by element”,选择C为青色的选项,即可更换蛋白条带颜色。(当然也可以更换为其他颜色)。

-叁-
设置操作对象。为了方便后续操作,我们将配体及与其发生相互作用的关键氨基酸残基设置为不同对象。
首先按照下图顺序,将配体设置为“lig”对象。

随后同样的方法,将氨基酸残基设置为“res”,为了方便操作,我们这里使用命令行操作。主要包括与配体发生相互作用的氨基酸Glu590、Met592、Asp668、Phe589、Phe669和Tyr591等。
输入以下命令“select res,resi 590 592 668 589 669 591”,按下回车确认即完成操作。可以看到右侧窗口出现了lig和res的项目,表示设置配体及氨基酸残基对象完成。

-肆-
隐藏不需要的部分,添加标签。依次输入以下命令,可根据个人情况进行更改。

单击Viewer界面右侧菜单所示对象res文本右侧的“L”,选择”residues”,即可为氨基酸残基添加标签。

-伍-
绘制蛋白-小分子结合作用力。由于小分子配体与氨基酸Phe589、Phe669形成π-π堆积作用,因此,我们以Phe589为例绘制π-π堆积作用。
首先我们确保选择(Selecting)模式为Atoms状态(更改方式位于Pymol软件的右下方,鼠标单击Selecting后面的选项即可更改),此时,鼠标单击选择的便不是残基而是单个原子,方便我们创建几何中心球。
依次点击Phe589残基苯环上的六个原子,输入命令“com sale,object=p1”,即可生成名称为p1的质心球对象;同样的方法,我们选中配体上咪唑环上的五个原子,输入命令“comsale,object=p2”,以及Phe669残基苯环上的六个原子,输入命令“comsale,object=p3”,即可生成名称为p2和p3的质心球对象。

随后,我们对小球的显示样式进行更改,输入以下命令。

所有的质心球创建完成后,开始绘制分子间作用力。点击Pymol菜单中的Wizard-Measurement,然后点击需要连接的小球/原子,两两一组,便可画出虚线,该虚线同样作为对象(如measure01)可以在右侧菜单中调整参数,同样的办法绘制氢键作用和阳离子- π作用,连接完成后点击右侧菜单的“done”即可退出连接模式。
-陆-
调整图像。为了使图片达到出版要求,我们需要对图像进行进一步修饰。首先对作用力虚线进行修饰,然后显示蛋白图。按以下命令进行操作。

我们还可以点击Pymol软件上方的菜单栏Setting-Cartoon,然后确保RoundHelices、FancyHelices、FlatSheets和FancySheets四个选项中打勾,对蛋白Cartoon图像进一步微调。
此外,我们可以移动label的位置。首先设置Mouse Mode为Editing模式,按住Ctrl键,用鼠标左键或右键拖动label。最后得到下图。

最后,为了得到更高品质的图像,可以在空白处点击鼠标右键,选择Ray即可进行图片光线渲染。另外,Pymol中的字体标注不是很好看,因此我们需要后期导入PS或者PPT里进一步处理,这时,为了方便标注,我们可以存一张带标签的和一张不带标签的图像,方便后期修改。
这时,我们可以输入命令“hide labels”和“show labels”来隐藏/显示标签。同样的方法可通过命令“show cartoon”和“hide cartoon”来显示/隐藏蛋白条带。可以通过按住鼠标右键拖动的方法来调节视角大小,通过按住鼠标左键拖动的方法转动视角。以此来获得更好的图像。
通过输入以下命令来导出图像。

下图中为导出的带标签的图像(左)和不带标签的图像(右),待所有需要的图像均保存完成后,再进行后期处理。
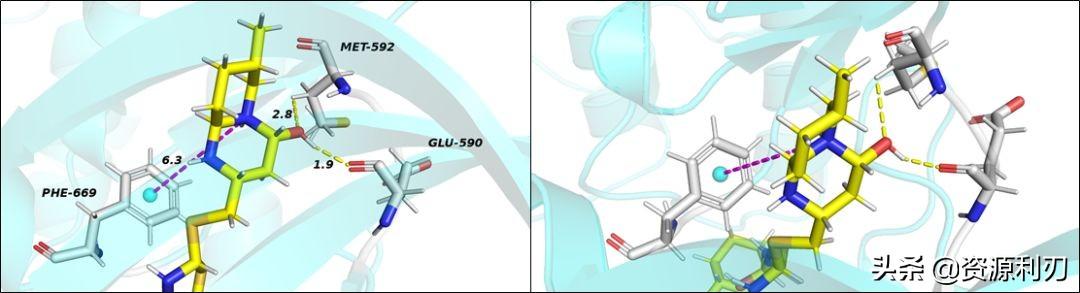
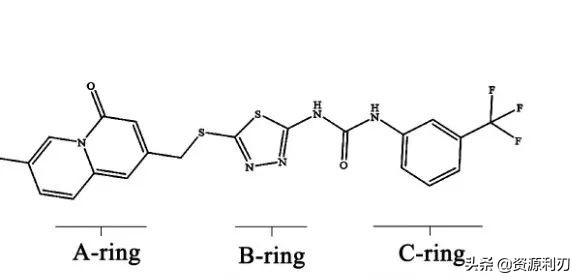
另外,由于这里小分子较大,一张图会显得很杂,利刃君这里将小分子分为3个片段:A-ring、B-ring和C-ring三个片段,来使蛋白-小分子的结合方式展示的更加清晰。
随后,熟悉PS的可以将图片导入到PS中进行图像拼合和文字添加以及排版,不熟悉PS的也可以用PPT进行处理。(PS方便我们进行图像大小设置,以达到杂志社的要求)这样就完成了最后的图片啦~

注:由于时间关系,本次教程中疏水作用未进行绘制,大家可以以文中同样的方法进行添加。
资源获取方式:
1.点击头像,添加关注;
2.点击头像,私信关键词:035(注意关键词不要多字少字,否则后台无法识别)
就可获取 Pymol软件的center_of_mass.py插件(附带安装教程)啦~
,免责声明:本文仅代表文章作者的个人观点,与本站无关。其原创性、真实性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容文字的真实性、完整性和原创性本站不作任何保证或承诺,请读者仅作参考,并自行核实相关内容。文章投诉邮箱:anhduc.ph@yahoo.com






